class: center, middle, inverse, title-slide .title[ # Estadística Multivariada ] .subtitle[ ## ⚔<br/> Clase 3. Exploración Multivariada ] .author[ ### Javier Rodríguez Barrios ] .institute[ ### Maestría en Ecología y Biodiversidad ] .date[ ### actualizado: 2024-04-17 ] --- class: inverse, center, middle # Empezamos --- # Descarga de datos Descargar la base de datos **Insectos1.csv** de la página [Github](https://github.com/Javier-2712/Multivariados.git): ```r remotes::install_github("yihui/xaringan") ``` -- - Seguir los procedimientos de las clases anteriores, para cargar los archivos **`.csv`** y **`.html`** en el directorio de trabajo. -- - Se puede hacer uso de la bibliografía recomendada por el docente. .pull[  ] --- ## Cargar la base de datos ```r # Base de datos datos<-read.csv2("Insectos.csv",row.names=1) head(datos) # Encabezado de la base "datos" ``` ``` # cuenca pH temp Efem Plec Tric Dipt Cole Ab # 1 cuen1 6.8 17.4 26 4 9 30 3 72 # 4 cuen1 7.3 16.8 17 6 9 25 1 58 # 11 cuen1 5.6 16.0 9 3 28 24 3 67 # 13 cuen1 6.3 17.8 2 3 25 21 6 57 # 19 cuen1 5.6 18.2 6 4 24 12 13 59 # 3 cuen2 6.3 17.0 7 2 25 10 1 45 ``` --- ## Estructura de la base de datos ```r str(datos) # estructura de la base "datos" ``` ``` # 'data.frame': 20 obs. of 9 variables: # $ cuenca: chr "cuen1" "cuen1" "cuen1" "cuen1" ... # $ pH : num 6.8 7.3 5.6 6.3 5.6 6.3 7.5 7 7 5.7 ... # $ temp : num 17.4 16.8 16 17.8 18.2 17 16.8 18.2 19.8 15.3 ... # $ Efem : int 26 17 9 2 6 7 19 12 13 5 ... # $ Plec : int 4 6 3 3 4 2 3 5 6 0 ... # $ Tric : int 9 9 28 25 24 25 12 23 9 32 ... # $ Dipt : int 30 25 24 21 12 10 12 9 0 11 ... # $ Cole : int 3 1 3 6 13 1 3 4 15 8 ... # $ Ab : int 72 58 67 57 59 45 49 53 43 56 ... ``` --- ### Librerías Requeridas ```r library(lattice) # No se requiere instalar library(ellipse) require(SciViews) library(plotrix) require(stats) # No se requiere instalar library(corrplot) ``` ### Estructura de la base de datos ```r str(datos) ``` ``` ## 'data.frame': 20 obs. of 9 variables: ## $ cuenca: chr "cuen1" "cuen1" "cuen1" "cuen1" ... ## $ pH : num 6.8 7.3 5.6 6.3 5.6 6.3 7.5 7 7 5.7 ... ## $ temp : num 17.4 16.8 16 17.8 18.2 17 16.8 18.2 19.8 15.3 ... ## $ Efem : int 26 17 9 2 6 7 19 12 13 5 ... ## $ Plec : int 4 6 3 3 4 2 3 5 6 0 ... ## $ Tric : int 9 9 28 25 24 25 12 23 9 32 ... ## $ Dipt : int 30 25 24 21 12 10 12 9 0 11 ... ## $ Cole : int 3 1 3 6 13 1 3 4 15 8 ... ## $ Ab : int 72 58 67 57 59 45 49 53 43 56 ... ``` ```r datos$cuenca = as.factor(datos$cuenca) summary(datos) ``` ``` ## cuenca pH temp Efem Plec ## cuen1:5 Min. :5.50 Min. :15.00 Min. : 2.00 Min. :0.00 ## cuen2:5 1st Qu.:6.00 1st Qu.:15.95 1st Qu.: 6.00 1st Qu.:3.00 ## cuen3:5 Median :6.50 Median :17.05 Median :12.50 Median :4.00 ## cuen4:5 Mean :6.48 Mean :16.99 Mean :13.75 Mean :3.85 ## 3rd Qu.:7.00 3rd Qu.:17.88 3rd Qu.:19.00 3rd Qu.:5.00 ## Max. :7.50 Max. :19.80 Max. :28.00 Max. :8.00 ## Tric Dipt Cole Ab ## Min. : 7.00 Min. : 0.00 Min. : 1.00 Min. : 43.00 ## 1st Qu.:15.00 1st Qu.:12.00 1st Qu.: 3.00 1st Qu.: 56.75 ## Median :22.00 Median :24.50 Median : 6.00 Median : 72.50 ## Mean :20.95 Mean :22.15 Mean : 7.70 Mean : 68.40 ## 3rd Qu.:25.00 3rd Qu.:30.00 3rd Qu.:12.25 3rd Qu.: 76.50 ## Max. :37.00 Max. :39.00 Max. :21.00 Max. :104.00 ``` --- class: inverse, center, middle # Exploración gráfica --- ### Propósito de la exploración <table> <thead> <tr> <th style="text-align:left;"> </th> <th style="text-align:left;"> cuenca </th> <th style="text-align:right;"> pH </th> <th style="text-align:right;"> temp </th> <th style="text-align:right;"> Efem </th> <th style="text-align:right;"> Plec </th> <th style="text-align:right;"> Tric </th> <th style="text-align:right;"> Dipt </th> <th style="text-align:right;"> Cole </th> <th style="text-align:right;"> Ab </th> </tr> </thead> <tbody> <tr> <td style="text-align:left;"> 1 </td> <td style="text-align:left;"> cuen1 </td> <td style="text-align:right;"> 6.8 </td> <td style="text-align:right;"> 17.4 </td> <td style="text-align:right;"> 26 </td> <td style="text-align:right;"> 4 </td> <td style="text-align:right;"> 9 </td> <td style="text-align:right;"> 30 </td> <td style="text-align:right;"> 3 </td> <td style="text-align:right;"> 72 </td> </tr> <tr> <td style="text-align:left;"> 4 </td> <td style="text-align:left;"> cuen1 </td> <td style="text-align:right;"> 7.3 </td> <td style="text-align:right;"> 16.8 </td> <td style="text-align:right;"> 17 </td> <td style="text-align:right;"> 6 </td> <td style="text-align:right;"> 9 </td> <td style="text-align:right;"> 25 </td> <td style="text-align:right;"> 1 </td> <td style="text-align:right;"> 58 </td> </tr> <tr> <td style="text-align:left;"> 11 </td> <td style="text-align:left;"> cuen1 </td> <td style="text-align:right;"> 5.6 </td> <td style="text-align:right;"> 16.0 </td> <td style="text-align:right;"> 9 </td> <td style="text-align:right;"> 3 </td> <td style="text-align:right;"> 28 </td> <td style="text-align:right;"> 24 </td> <td style="text-align:right;"> 3 </td> <td style="text-align:right;"> 67 </td> </tr> <tr> <td style="text-align:left;"> 13 </td> <td style="text-align:left;"> cuen1 </td> <td style="text-align:right;"> 6.3 </td> <td style="text-align:right;"> 17.8 </td> <td style="text-align:right;"> 2 </td> <td style="text-align:right;"> 3 </td> <td style="text-align:right;"> 25 </td> <td style="text-align:right;"> 21 </td> <td style="text-align:right;"> 6 </td> <td style="text-align:right;"> 57 </td> </tr> <tr> <td style="text-align:left;"> 19 </td> <td style="text-align:left;"> cuen1 </td> <td style="text-align:right;"> 5.6 </td> <td style="text-align:right;"> 18.2 </td> <td style="text-align:right;"> 6 </td> <td style="text-align:right;"> 4 </td> <td style="text-align:right;"> 24 </td> <td style="text-align:right;"> 12 </td> <td style="text-align:right;"> 13 </td> <td style="text-align:right;"> 59 </td> </tr> <tr> <td style="text-align:left;"> 3 </td> <td style="text-align:left;"> cuen2 </td> <td style="text-align:right;"> 6.3 </td> <td style="text-align:right;"> 17.0 </td> <td style="text-align:right;"> 7 </td> <td style="text-align:right;"> 2 </td> <td style="text-align:right;"> 25 </td> <td style="text-align:right;"> 10 </td> <td style="text-align:right;"> 1 </td> <td style="text-align:right;"> 45 </td> </tr> </tbody> </table> -- **Objetivo 1**. **Relaciones** o **Correlaciones** entre variables (Ej. Bióticas vs. Ambientales) **Exploratorios**. Figuras de elipses, de dispersión, ... --- ### Propósito de la exploración ``` *## cuenca pH temp Efem Plec Tric Dipt Cole Ab ## 1 cuen1 6.8 17.4 26 4 9 30 3 72 ## 4 cuen1 7.3 16.8 17 6 9 25 1 58 ## 11 cuen1 5.6 16.0 9 3 28 24 3 67 ## 13 cuen1 6.3 17.8 2 3 25 21 6 57 ## 19 cuen1 5.6 18.2 6 4 24 12 13 59 ## 3 cuen2 6.3 17.0 7 2 25 10 1 45 ``` -- **Objetivo 2**. **Diferencias** de las variables entre grupos de observaciones (Factor) **Exploratorios**. Figuras de **cajas y bigotes**, de promedios y desviaciones, ... --- # Videos de clases - Exploratorios Video de la clase con opciones de elipses .pull-left[ [1. Cargar base de datos en RStudio](https://web.microsoftstream.com/video/784dda7e-d129-4d84-b2fd-e3831c3006b2) [2. Figuras de pares y de elipses](https://web.microsoftstream.com/video/867c9668-6f00-4f5c-afd0-2ee945913fe2) [3. Figuras de pares avanzadas](https://web.microsoftstream.com/video/b2c6b123-167c-416d-8e5c-b6024459d663) [4. Figura Trivariada - Coplot](https://web.microsoftstream.com/video/3db58f9e-b5f2-4b45-b760-d827d511788f) [5. Histogramas](https://web.microsoftstream.com/video/f74cb6da-5d0f-4c49-8492-332b0f4e231b) ] .pull-right[  ] --- class: inverse, center, middle # Elipses ## Correlaciones entre variables --- ## 1.) Elipses con colores Las elipses permiten evaluar relaciones entre múltiples variables. El Paquete **Corrplot** es el que permite realizar las opciones gráficas de elipses a color, ingresar a este enlace: [corrplot](http://www.sthda.com/english/wiki/visualize-correlation-matrix-using-correlogram) O ete otro enlace a **corrplot**: [corrplot](https://cran.r-project.org/web/packages/corrplot/vignettes/corrplot-intro.html) -- A continuación se presenta la estructura básica de estas figuras ````markdown # Matriz de Correlación (M) M <- cor(datos[,2:9]) M ``` -- ````markdown # Figura de correlaciones con elipses corrplot(M, method = "ellipse") ``` --- ## 1.) Elipses con colores ```r # Elipses con colores (Obj1 - Reación v.biol vs. v.f.q) M <- cor(datos[,2:9]) # Matriz de Correlación (M) corrplot(M, method = "ellipse") # Figura de correlaciones con elipses ``` <!-- --> --- ## 1.) Elipses con colores (*Cont.*) ### Elipses con transformación de las variables (log10) ```r corrplot(log10(M+1), method = "ellipse") # Figura de correlaciones con elipses ``` <!-- --> --- ## 1.) Elipses con colores (*Cont.*) El Paquete **Corrplot** es el que permite realizar diferentes opciones gráficas de elipses a color, como las del siguiente enlace: [corrplot](http://www.sthda.com/english/wiki/visualize-correlation-matrix-using-correlogram) -- ### Otras opciones de elipses ````markdown # Figura de correlaciones con círculos corrplot(M, method = "circle") corrplot.mixed(M, upper="ellipse") ``` -- **`M`** es la matriz de correlación de pearson **`circle`** es la figura con la matriz de circulos **`corrplot.mixed`** combina elipses y circulos **`upper=ellipse`** coloca las elipses encima --- ### Otras opciones de elipses .pull-left[ Figura de correlaciones con **circulos** **Azul** representa relaciones positivas y **rojo** a las negativas ```r *corrplot(M, method = "circle") ``` ] .pull-right[ Figura generada 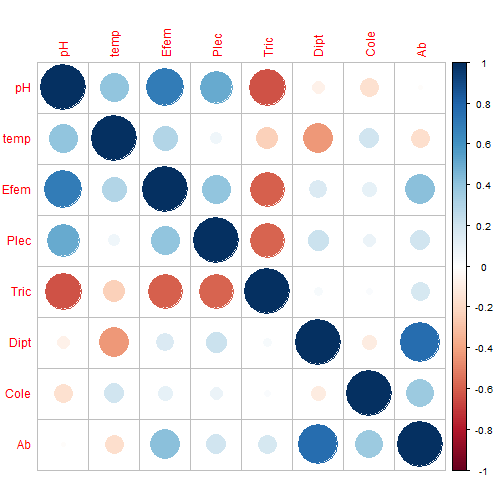<!-- --> ] --- class:inverse .pull-left[ Figura de **elipses** con coeficientes de correlación de pearson. **Azul** representa relaciones positivas y **rojo** a las negativas ```r *corrplot.mixed(M, upper="ellipse") ``` ] .pull-right[ Figura generada 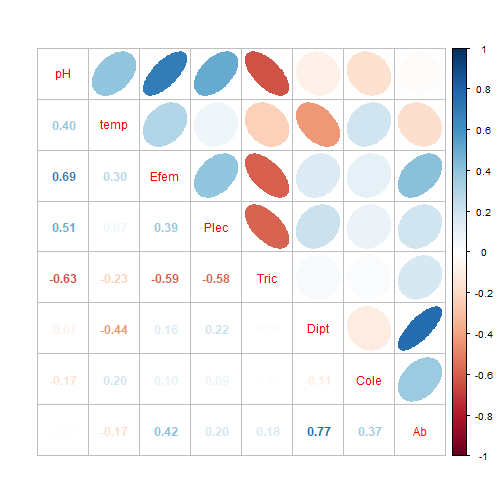<!-- --> ] -- Algunas modificaciones de **ggplot2** permiten hacer figuras de elipses, pueden explorarse en el siguiente enlace: [paquete ggpairs](https://r-graph-gallery.com/97-correlation-ellipses.html) --- ## Fuente de esta figura [paquete ggpairs](https://r-graph-gallery.com/97-correlation-ellipses.html) <div> <p style = 'text-align:center;'> <img src="https://r-graph-gallery.com/97-correlation-ellipses_files/figure-html/thecode-1.png" alt="JuveYell" width="500px"> </p> </div> --- # 1) Figuras de elipses .left-column[ ## Resumen ## Figuras de Elipses ] .right-column[ - Permiten visualizar **relaciones** lineales positivas o negativas entre parejas de variables. - Permiten realizar **realaciones y correlaciones** entre variables biológicas. - Permiten **correlacionar** a variables biológicas y ambientales. ] <div> <p style = 'text-align:center;'> <img src="https://r-graph-gallery.com/97-correlation-ellipses_files/figure-html/thecode-1.png" alt="JuveYell" width="300px"> </p> </div> #---- --- class: inverse, center, middle # Relaciones (Regresiones) ## Figuras de dispersión --- ## 2.) Figuras de dispersión Preden relaciones de **dos variables** seleccionadas de aquellas que en las figuras de elipses presentaron la mayor relación. -- El Paquete **`lattice`** es uno de los que permite realizar las opciones gráficas de dispersión, ingresar a este enlace: [lattice](http://www.sthda.com/english/wiki/lattice-graphs) -- El Paquete **`ggplot2`** presenta opciones más estéticas y robustas para estas y muchas más figuras, ingresar a este enlace: [Enlace1](https://r-charts.com/es/correlacion/grafico-dispersion-ggplot2/) [Enlace2](https://soka.gitlab.io/blog/post/2019-04-25-r-ggplot2-scatterplot/) -- El Paquete **`ggppairs`** presenta opciones para relacionar diferentes variables: [ggpairs in R](https://www.r-bloggers.com/2022/11/ggpairs-in-r/?utm_source=phpList&utm_medium=email&utm_campaign=R-bloggers-daily&utm_content=HTML) --- A continuación se presenta la estructura básica de estas figuras ````markdown # Figura de dispersión en `ggplot2` ggplot(datos,aes(x = pH,y = Efem)) + geom_point() + geom_smooth() ``` -- ````markdown # Figura de dispersión con regresiones (`smooth`) ggplot(datos,aes(x = pH,y = Efem)) + geom_point(aes(color = cuenca), size = 3) + geom_smooth(method= "lm") ``` --- ### 1.) Elipses con colores (Ver relación Efm vs. pH) ```r # Elipses con colores (Obj1 - Reación v.biol vs. v.f.q) M <- cor(datos[,2:9]) # Matriz de Correlación (M) corrplot(M, method = "ellipse") # Figura de correlaciones con elipses ``` <!-- --> --- # Librerías Requeridas ```r library(lattice) library(tidyverse) library(ggplot2) ``` **Nota** La base de datos **`Insectos.csv`** debe cargarse desde el directorio de trabajo. --- ## 2.) Regresiones lineales (Esquema básico) ```r plot(Efem~Plec,col=as.integer(cuenca),data=datos) legend(0,25,legend=levels(datos$cuenca),pch=19,col=1:4,cex=0.8) lines(abline(lm(datos$Efem~datos$Plec),lwd=2,col=2, lty=2)) ``` <!-- --> --- ## 3.) Regresiones lineales (Esquema ggplot2) ```r ggplot(datos,aes(x = pH,y = Efem)) + geom_point(aes(color = cuenca), size = 3) + geom_smooth(method= "lm") ``` <!-- --> --- ## 4.) Regresiones suavizadas (Loess) ```r ggplot(datos,aes(x = pH, y = Efem)) + geom_point(aes(color = cuenca), size = 3) + geom_smooth() ``` <!-- --> --- ## 5.) Regresiones ```r ggplot(datos,aes(x = pH, y = Efem)) + geom_point(aes(color = cuenca), size = 3) + geom_smooth(se = FALSE, span = 0.4) ``` <!-- --> #---- --- class: inverse, center, middle # 3.) Figuras de Cajas y Bigotes ## Figura de variariaciones o diferencias --- ## 3.) Figuras de cajas y bigotes Se valoran las diferencias en los patrones de las variables entre grupos (definidos por uno o más factores). -- El Paquete **`lattice`** es uno de los que permite realizar las opciones gráficas de cajas, ingresar a este enlace: [How to make a boxplot in R](https://www.r-bloggers.com/2022/04/how-to-make-a-boxplot-in-r/?utm_source=phpList&utm_medium=email&utm_campaign=R-bloggers-daily&utm_content=HTML) El Paquete **`ggplot2`** presenta opciones más estéticas y robustas para estas y muchas más figuras, ingresar a este enlace: [Enlace1](https://r-charts.com/es/correlacion/grafico-dispersion-ggplot2/) [Enlace2](https://soka.gitlab.io/blog/post/2019-04-25-r-ggplot2-scatterplot/) -- A continuación se presenta la estructura básica de estas figuras ````markdown # Figura de cajas en `ggplot2` ggplot(datos, aes(x=cuenca, y=Ab)) + geom_boxplot() ``` -- ````markdown # Figura de cajas con otras opciones (`fill`) ggplot(datos, aes(x=cuenca, y=Ab)) + geom_boxplot(col="Blue", fill="orange") + labs(x="Cuencas", y= "Abundancia de Insectos") + theme(axis.text= element_text(size=14)) ``` --- # Librerías Requeridas ```r library(tidyverse) library(ggplot2) library(reshape2) library(gridExtra) library(gtable) library(grid) library(ggforce) ``` **Nota** La base de datos **`Insectos.csv`** debe cargarse con tidyverse. --- #### Cargar base de datos ```r # Base de datos datos<-read_csv2("Insectos.csv") # Formato *Tidyr head(datos) # Encabezado de la base "datos" ``` ``` # # A tibble: 6 × 10 # quebrada cuenca pH temp Efem Plec Tric Dipt Cole Ab # <dbl> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> # 1 1 cuen1 6.8 17.4 26 4 9 30 3 72 # 2 4 cuen1 7.3 16.8 17 6 9 25 1 58 # 3 11 cuen1 5.6 16 9 3 28 24 3 67 # 4 13 cuen1 6.3 17.8 2 3 25 21 6 57 # 5 19 cuen1 5.6 18.2 6 4 24 12 13 59 # 6 3 cuen2 6.3 17 7 2 25 10 1 45 ``` ```r str(datos) # estructura de la base "datos" ``` ``` # spc_tbl_ [20 × 10] (S3: spec_tbl_df/tbl_df/tbl/data.frame) # $ quebrada: num [1:20] 1 4 11 13 19 3 10 15 16 17 ... # $ cuenca : chr [1:20] "cuen1" "cuen1" "cuen1" "cuen1" ... # $ pH : num [1:20] 6.8 7.3 5.6 6.3 5.6 6.3 7.5 7 7 5.7 ... # $ temp : num [1:20] 17.4 16.8 16 17.8 18.2 17 16.8 18.2 19.8 15.3 ... # $ Efem : num [1:20] 26 17 9 2 6 7 19 12 13 5 ... # $ Plec : num [1:20] 4 6 3 3 4 2 3 5 6 0 ... # $ Tric : num [1:20] 9 9 28 25 24 25 12 23 9 32 ... # $ Dipt : num [1:20] 30 25 24 21 12 10 12 9 0 11 ... # $ Cole : num [1:20] 3 1 3 6 13 1 3 4 15 8 ... # $ Ab : num [1:20] 72 58 67 57 59 45 49 53 43 56 ... # - attr(*, "spec")= # .. cols( # .. quebrada = col_double(), # .. cuenca = col_character(), # .. pH = col_double(), # .. temp = col_double(), # .. Efem = col_double(), # .. Plec = col_double(), # .. Tric = col_double(), # .. Dipt = col_double(), # .. Cole = col_double(), # .. Ab = col_double() # .. ) # - attr(*, "problems")=<externalptr> ``` --- ## 1.) Figura de cajas - 1 variable ```r ggplot(datos, aes(x=cuenca, y=Ab)) + geom_boxplot() ``` <!-- --> --- ## 2.) Ajuste de colores ```r ggplot(datos, aes(x=cuenca, y=Ab)) + geom_boxplot(col="Blue", fill="orange") + labs(x="Cuencas", y= "Abundancia de Insectos") + theme(axis.text= element_text(size=14)) ``` <!-- --> --- ## 3) Multiples variables ```r ggplot(melt(datos[,c(2,5:10)]), aes(x=variable, y=value)) + geom_boxplot(aes(fill=cuenca)) + labs(x="",y="Abundancia") ``` <!-- --> --- ## 3) Otra forma - multiples variables ```r ggplot(melt(datos[,c(2,5:10)]), aes(x=variable, y=value)) + geom_boxplot(aes(fill=cuenca)) + labs(x="",y="Abundancia") + facet_wrap(~ variable,scales="free") + theme_bw() ``` <!-- --> --- class: inverse, center, middle # 4) Figuras con estadísticos (promedios, errores, ...) ## Figura de variariaciones o diferencias --- ## 4) Figuras con estadísticos Similar a lo definido en las figuras de cajas y bigotes, aquí se valoran las diferencias en los patrones de las variables entre grupos (definidos por uno o más factores). -- El Paquete **`ggplot2`** es uno de los que permite realizar las opciones gráficas de barras con estadísticos, ingresar a este enlace: [**Exploratory Data Analysis with ggplot**](https://rstudio-pubs-static.s3.amazonaws.com/355851_52530dca7f8a4355aad5f5ba46c88b42.html) Existen otros enlaces en los que se puede encontrar información complementaria para figuras de barras, como los siguientes [**ggplot2 barplots**](https://rstudio-pubs-static.s3.amazonaws.com/355851_52530dca7f8a4355aad5f5ba46c88b42.html) [**Stunning Bar Charts**](https://appsilon.com/ggplot2-bar-charts/) --- # Librerías Requeridas ```r library(tidyverse) library(ggplot2) library(grid) library(ggforce) library(GGally) library(gganimate) ``` **Nota** La base de datos **`Insectos.csv`** debe cargarse con tidyverse. --- A continuación se presenta la estructura básica de estas figuras 1) Cálculo de los estadísticos a tabular y graficar ````markdown datos_resum <- datos %>% # Base de datos resumida group_by(cuenca) %>% # Factor o variable agrupadora summarise(datos.m = mean(Ab), # Media de cada grupo del factor datos.de = sd(Ab), # Desviacioes estándar de cada grupo datos.var = var(Ab), # Varianzas de cada grupo n.Ab = n(), # Tamaño de cada grupo datos.ee = sd(Ab)/sqrt(n())) # Error estándar de cada grupo datos_resum ``` -- 2) Generación de las figuras ````markdown ggplot(datos_resum, aes(cuenca, datos.m, dev.off())) + geom_bar(stat="identity", col="blue", fill="lightblue") + geom_errorbar(aes(ymin=datos.m-datos.ee, ymax=datos.m+datos.ee),width=0.2) ``` --- #### Cargar base de datos ```r # Base de datos datos<-read_csv2("Insectos.csv") # Formato *Tidyr head(datos) # Encabezado de la base "datos" ``` ``` # # A tibble: 6 × 10 # quebrada cuenca pH temp Efem Plec Tric Dipt Cole Ab # <dbl> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> # 1 1 cuen1 6.8 17.4 26 4 9 30 3 72 # 2 4 cuen1 7.3 16.8 17 6 9 25 1 58 # 3 11 cuen1 5.6 16 9 3 28 24 3 67 # 4 13 cuen1 6.3 17.8 2 3 25 21 6 57 # 5 19 cuen1 5.6 18.2 6 4 24 12 13 59 # 6 3 cuen2 6.3 17 7 2 25 10 1 45 ``` ```r str(datos) # estructura de la base "datos" ``` ``` # spc_tbl_ [20 × 10] (S3: spec_tbl_df/tbl_df/tbl/data.frame) # $ quebrada: num [1:20] 1 4 11 13 19 3 10 15 16 17 ... # $ cuenca : chr [1:20] "cuen1" "cuen1" "cuen1" "cuen1" ... # $ pH : num [1:20] 6.8 7.3 5.6 6.3 5.6 6.3 7.5 7 7 5.7 ... # $ temp : num [1:20] 17.4 16.8 16 17.8 18.2 17 16.8 18.2 19.8 15.3 ... # $ Efem : num [1:20] 26 17 9 2 6 7 19 12 13 5 ... # $ Plec : num [1:20] 4 6 3 3 4 2 3 5 6 0 ... # $ Tric : num [1:20] 9 9 28 25 24 25 12 23 9 32 ... # $ Dipt : num [1:20] 30 25 24 21 12 10 12 9 0 11 ... # $ Cole : num [1:20] 3 1 3 6 13 1 3 4 15 8 ... # $ Ab : num [1:20] 72 58 67 57 59 45 49 53 43 56 ... # - attr(*, "spec")= # .. cols( # .. quebrada = col_double(), # .. cuenca = col_character(), # .. pH = col_double(), # .. temp = col_double(), # .. Efem = col_double(), # .. Plec = col_double(), # .. Tric = col_double(), # .. Dipt = col_double(), # .. Cole = col_double(), # .. Ab = col_double() # .. ) # - attr(*, "problems")=<externalptr> ``` --- ## 1.) Estadísticos "datos_resum" ```r # Resumen estadístico "datos_resum" datos_resum <- datos %>% # Base de datos resumida group_by(cuenca) %>% # Factor o variable agrupadora summarise(datos.m = mean(Ab), # Media de cada grupo del factor datos.de = sd(Ab), # Desviacioes estándar de cada grupo datos.var = var(Ab), # Varianzas de cada grupo n.Ab = n(), # Tamaño de cada grupo datos.ee = sd(Ab)/sqrt(n())) # Error estándar de cada grupo datos_resum ``` ``` ## # A tibble: 4 × 6 ## cuenca datos.m datos.de datos.var n.Ab datos.ee ## <chr> <dbl> <dbl> <dbl> <int> <dbl> ## 1 cuen1 62.6 6.58 43.3 5 2.94 ## 2 cuen2 49.2 5.40 29.2 5 2.42 ## 3 cuen3 73.8 1.30 1.7 5 0.583 ## 4 cuen4 88 10.1 102. 5 4.53 ``` --- ## 2.) Elementos de la figura "DatosPlot" ```r # Figura de promedios y errores estándar DatosPlot<- ggplot(datos_resum, aes(cuenca, datos.m, dev.off())) + geom_bar(stat="identity", col="blue", fill="lightblue") + geom_errorbar(aes(ymin=datos.m-datos.ee, ymax=datos.m+datos.ee),width=0.2) ``` --- ## 3.) Impresión de la figura "print" ```r # Imprimir la figura de promedios con errores estándar print (DatosPlot + labs(y="Abundancia ± e.e.", x = "cuenca") + scale_fill_manual(values= c("#A1D5D5")) + theme_classic()) ``` <!-- --> --- ***Nota:*** Para las figuras de **Cajas y Bigotes**, el comando `stat_boxplot(geom = "errorbar",...)` permite realizar gráficas sin necesidad de extraer algunos estadísticos previamente. ```r # Opción de cajas y bigotes con errores estándar f <- ggplot(datos, aes(x=cuenca, y= Ab, fill= cuenca)) + stat_boxplot(geom = "errorbar",width = 0.05) + geom_boxplot(width = 0.4, notchwidth = 0.9, outlier.colour="red", outlier.fill="red", outlier.size=3, outlier.alpha = 0.2) + theme_classic() + scale_fill_manual(values=c('#fc8d59','#ffffbf','#99d594','#377eb8')) + labs(x = "Cuencas", y = "Abundancia ± e.e.")+ scale_y_continuous(limits = c(40,90)) ``` --- ## 4.) Impresión de la figura "print" ```r print(f) ``` <!-- --> --- <b style='color:#1f78b4;'> ### Base de datos con múltiples factores </b> #### Cargar base de datos ```r # Base de datos multifactorial (insectos1) datos1<-read_csv2("Insectos2.csv") # Formato *xlsx head(datos1) # Encabezado ``` ``` # # A tibble: 6 × 6 # No Muestreo GF Lluvia Ab Biom # <dbl> <chr> <chr> <chr> <dbl> <dbl> # 1 1 M1 C-F P1 98 56.0 # 2 2 M2 C-F P1 198 52.7 # 3 3 M3 C-F P2 45 11.4 # 4 4 M4 C-F P2 51 25.3 # 5 5 M5 C-F P2 3 0.36 # 6 6 M6 C-F P2 69 23.6 ``` --- ## 1.) Estadísticos "datos_resum" ```r # Resumen estadístico "datos_resum" datos_resum <- datos1 %>% # Base de datos resumida group_by(Lluvia,GF) %>% # Factor o variable agrupadora summarise(datos.m = mean(Biom), # Media de cada grupo del factor datos.de = sd(Biom), # Desviacioes estándar de cada grupo datos.var = var(Biom), # Varianzas de cada grupo n.Biom = n(), # Tamaño de cada grupo datos.ee = sd(Biom)/sqrt(n())) # Error estándar de cada grupo datos_resum ``` ``` # # A tibble: 10 × 7 # # Groups: Lluvia [2] # Lluvia GF datos.m datos.de datos.var n.Biom datos.ee # <chr> <chr> <dbl> <dbl> <dbl> <int> <dbl> # 1 P1 C-F 37.0 30.0 902. 3 17.3 # 2 P1 C-R 31.7 32.1 1029. 3 18.5 # 3 P1 D 361. 120. 14411. 3 69.3 # 4 P1 R 53.5 69.8 4873. 3 40.3 # 5 P1 T 190. 296. 87533. 3 171. # 6 P2 C-F 14.4 10.2 105. 5 4.58 # 7 P2 C-R 88.5 115. 13273. 5 51.5 # 8 P2 D 176. 94.9 9010. 5 42.4 # 9 P2 R 21.9 12.9 165. 5 5.75 # 10 P2 T 151. 223. 49655. 5 99.7 ``` --- ## 2.) Elementos de las dos figuras "f1" y "f2" ```r # Figura 1 (f1) f1 = ggplot(datos_resum, aes(x=GF, y=datos.m, fill=Lluvia)) + geom_bar(stat="identity", color="black", position=position_dodge()) + geom_errorbar(aes(ymin=datos.m, ymax=datos.m+datos.de), width=.2, position=position_dodge(.9)) # f2: Otro formato de figura bifactorial - theme_classic f2 = f1+labs(title="Biomasa por GF", x="Grupos Funcionales - GF", y = "Biomasa")+ theme_classic() + scale_fill_manual(values=c('#E69F00','#999999')) ``` --- ## 3.) Impresión de las figuras "grid.arrange" ```r # Impresión de un panel con las dos figuras (p1 y p2) grid.arrange (f1, f2, ncol=2) ``` <!-- --> --- ```r # Inserción de las figuras en columna (figuras p1 y p2) f3 <- ggplotGrob(f1) f4 <- ggplotGrob(f2) g <- rbind(f3, f4, size="first") g$widths <- unit.pmax(f3$widths, f4$widths) grid.newpage() grid.draw(g) ``` <!-- --> --- <b style='color:#1f78b4;'> ## **parentesis** Figuras de dispersión animadas </b> [Enlace_de_figuras_animadas](https://spcanelon.notion.site/gganimate-How-to-Create-Plots-with-Beautiful-Animation-in-R-Datanovia-7193ba557c4345c3bbb6578b2b40e41f) #### Cargar base de datos ```r library(ggplot2) library(gganimate) theme_set(theme_bw()) # Tema o fondo de la figura por default ``` -- ## 2.) Elementos de la figura "p" ```r # Demo library(gapminder) p <- ggplot( gapminder, aes(x = gdpPercap, y=lifeExp, size = pop, colour = country) ) + geom_point(show.legend = FALSE, alpha = 0.7) + scale_color_viridis_d() + scale_size(range = c(2, 12)) + scale_x_log10() + labs(x = "GDP per capita", y = "Life expectancy") ``` --- ## 3) Impresión de la figura ```r p + transition_time(year) + labs(title = "Year: {frame_time}") ``` ```r p ``` <!-- -->